Optimización diagnóstica en selección embrionaria personalizada: secuenciación masiva versus masiva con polimorfismos de nucleótido único
Autores:
Liliana Berenice Ramírez Domínguez1, Victoria Stefani Varela Orozco2, Alexandra Bermudez Rodríguez3.
Resumen
El diagnóstico genético preimplantacional para aneuploidías (PGTA) mediante secuenciación de nueva generación (NGS) es ampliamente utilizado en reproducción asistida. Sin embargo, su sensibilidad para detectar alteraciones segmentales y mosaicismos puede ser limitada. La integración de polimorfismos de nucleótido único (SNP-array) podría optimizar la caracterización genética embrionaria. Por esta razón, el objetivo principal del presente estudio fue comparar la capacidad diagnóstica de secuenciación masiva combinado con polimorfismo de nucleótido único en la detección de aneuploidías, mosaicismos y alteraciones segmentales. Se realizó un estudio observacional, retrospectivo, realizado en embriones biopsiados en estadio de blastocisto. El análisis genético se realizó utilizando Ion Torrent™ (Thermo Fisher Scientific®) para secuenciación masiva y CytoScan HD (Thermo Fisher Scientific®) para SNP-array. Se compararon distribuciones diagnósticas mediante pruebas de Fisher exact y Chi-cuadrado.
El análisis estadístico se realizó con R v4.2.3. La cohorte de secuenciación masiva + polimorfismos de nucleótido único presentó una mayor detección de aneuploidías completas (57.1%), mosaicos (11.4%) y segmentales (7.1%) en comparación con secuenciación masiva. La discordancia diagnóstica global mostró tendencia a significancia (p=0.0592). Se propuso un algoritmo clínico preliminar para optimizar la selección embrionaria. Los resultados indicaron que la integración de SNP-array en el análisis genético mejora la resolución diagnóstica y permite una caracterización embrionaria más precisa. Estos hallazgos respaldan su aplicación dirigida en casos seleccionados, favoreciendo la selección personalizada de embriones en FIV.
Abstract
Preimplantation genetic testing for aneuploidy (PGTA) using next-generation sequencing (NGS) is widely employed in assisted reproductive technology (ART). However, its sensitivity for detecting segmental abnormalities and mosaicism may be limited. The integration of single nucleotide polymorphism arrays (SNP-array) could enhance embryo genetic characterization. Therefore, the main objective of this study was to compare the diagnostic performance of PGTA using next generation sequencing versus next generation sequencing combined with single nucleotide polimorphisms-array in detecting aneuploidies, mosaicism, and segmental alterations. We conducted an observational, retrospective study on blastocyst-stage biopsied embryos. Genetic analysis was performed using Ion Torrent™ (Thermo Fisher Scientific®) for next generation sequencing and CytoScan HD (Thermo Fisher Scientific®) for single nucleotide polimorphism. Diagnostic distributions were compared using Fisher’s exact and Chi-square tests. Statistical analysis was performed with R v4.2.3. The next generation sequencing + single nucleotide polimorphism cohort showed higher detection rates of complete aneuploidies (57.1%), mosaicism (11.4%), and segmental alterations (7.1%) compared to next generation sequencing alone. Global diagnostic discordance showed a trend towards significance (p = 0.0592). A preliminary clinical decision-making algorithm was proposed to optimize embryo selection. The results indicated that integrating single nucleotide polimorphisms into genetic analysis improves diagnostic resolution and enables more precise embryo characterization. These findings support its targeted application in selected cases, enhancing personalized embryo selection strategies in IVF.
Palabras clave
Diagnóstico genético preimplantacional, polimorfismo de un solo nucleótido, secuenciación de próxima generación, aneuploidía, fertilización in vitro
Key words
Preimplantation genetic testing, single nucleotide polimorphism, next generation sequencing, aneuploidy, in vitro fertilization
Introducción
La fertilización in vitro (FIV) ha revolucionado el tratamiento de la infertilidad; sin embargo, persiste el desafío de identificar los embriones con potencial genético para lograr un embarazo exitoso. La aneuploidía, definida como un número anormal de cromosomas, es una de las principales causas de fallos de implantación y pérdidas gestacionales tempranas 1. Para abordar este problema, se desarrolló la el diagnóstico genético preimplantacional para aneuploidías (PGT-A), que examina el complemento cromosómico de los embriones generados por reproducción asistida y permite seleccionar aquellos cromosómicamente normales (euploides) 2. Al transferir selectivamente embriones euploides, el diagnóstico genético preimplantacional para aneuploidía busca aumentar las tasas de implantación y nacidos vivos, y reducir los abortos espontáneos, optimizando la eficacia de los ciclos de FIV 3.
A lo largo de las últimas décadas, las estrategias de diagnóstico genético preimplantacional para aneuploidía han evolucionado notablemente. Inicialmente, técnicas de hibridación in situ fluorescente (FISH) evaluaban solo un número limitado de cromosomas, con sensibilidad reducida para detectar aneuploidías. Posteriormente, el cribado cromosómico completo fue posible gracias a tecnologías como los microarreglos genómicos y la secuenciación masiva o de nueva generación (NGS). En particular, los microarreglos de polimorfismo de nucleótido único (SNP array) surgieron como herramientas para detectar aneuploidías en todos los cromosomas 4
La secuenciación masiva (NGS) se ha posicionado como la plataforma predominante para diagnóstico genético preimplantacional para aneuploidía gracias a su alto rendimiento y capacidad para detectar mosaicismos y aneuploidías segmentarias. La secuenciación masiva ofrece ventajas significativas frente al SNP array, como mayor sensibilidad para identificar embriones mosaico e incluso un menor costo por análisis 5. Aunque los microarreglos permiten el tamizaje cromosómico completo, no cuantifica el grado de mosaicismo y puede omitir mosaicos de bajo porcentaje. Además, se han reportado discrepancias entre ambas plataformas al analizar embriones mosaico 6
Dado que cada plataforma aporta fortalezas distintas, resulta pertinente evaluar la integración de polimorfismos de nucleótido único en la práctica clínica del diagnóstico genetico preimplantacional. Comparar ambas metodologías en un contexto real permite determinar si su uso conjunto mejora la confiabilidad de la selección embrionaria, especialmente en la detección de mosaicismos y aneuploidías segmentarias. Estas anomalías sutiles son críticas para la selección personalizada de embriones, ya que pueden influir en la decisión de transferencia y en los resultados del tratamiento.
El presente trabajo representa la primera serie clínica mexicana conocida que explora estas metodologías en población real, con el objetivo de evaluar la detección de mosaicos, alteraciones segmentales y su posible impacto en la optimización de la selección embrionaria personalizada.
Objetivo general
Comparar la capacidad diagnóstica del test genético preimplantacional con secuenciación masiva versus secuenciación masiva con polimorfismo s de nucleótido único en la caracterización genética embrionaria.
Objetivos particulares: Comparar la distribución diagnóstica entre secuenciación masiva y polimorfismos de nucleótido único. Evaluar la discordancia diagnóstica mediante análisis inferencial. Proponer un algoritmo clínico preliminar para optimizar la selección embrionaria.
Material y Métodos
Estudio observacional, retrospectivo, comparativo y analítico, realizado en una cohorte de embriones humanos derivados de procedimientos de fertilización in vitro (FIV), evaluados mediante diagnóstico genético preimplantacional para aneuploidías (PGTA). Se incluyeron embriones en estadio de blastocisto (día 5-6 de cultivo), obtenidos de ciclos clínicos realizados entre enero de 2022 y marzo de 2025, sin restricciones de edad o indicación. Los embriones se agruparon según la técnica diagnóstica aplicada: secuenciación masiva (Next-Generation Sequencing, NGS). secuenciación masiva con polimorfismo s de nucleótido único. La biopsia de trofoectodermo se realizó mediante micromanipulación estándar y láser (ZILOS-tK®, Hamilton Thorne), extrayendo de 4 a 6 células por embrión. El análisis genético fue realizado por Igenomix México, utilizando las plataformas Ion Torrent™ (Thermo Fisher Scientific®, Waltham, MA, USA), basada en secuenciación por semiconductores, para la detección de aneuploidías cromosómicas y para SNP-array de alta densidad (CytoScan HD, Thermo Fisher Scientific®), para la detección de alteraciones segmentales, mosaicos y análisis de heterocigosidad.
Se compararon proporciones diagnósticas mediante la prueba de Fisher exact (para categorías específicas: euploide, aneuploide, mosaico, caótico, segmental) y la prueba de Chi-cuadrado (para análisis global de discordancia diagnóstica). Se consideró significancia estadística con p<0.05. El análisis se realizó en R v4.2.3 (R Foundation for Statistical Computing, Vienna, Austria), con las librerías tidyverse v2.0.0,stats v4.2.3, ggplot2 v3.4.2, epiR v2.0.50 y gmodels v2.18.1 para pruebas inferenciales y exploratorias.El protocolo fue aprobado por el Comité de Ética de la institución (registro CE-25-104). Se utilizó material embrionario de procedimientos clínicos con consentimiento informado, sin intervenciones experimentales adicionales.
Resultados
Se analizaron 33 ciclos con secuenciación masiva y 19 ciclos con secuenciación masiva más polimorfismos de nucleótido único, La Tabla 1 muestra las frecuencias diagnósticas observadas, destacando un aumento en la detección de mosaicos de bajo grado y alteraciones segmentales en el grupo secuenciación masiva más polimorfismos de nucleótido único.
La Tabla 1 muestra la distribución diagnóstica de los embriones analizados mediante secuenciación masiva y secuenciación masiva más polimorfismos de nucleótido único.
En el grupo NGS, el diagnóstico más frecuente fue aneuploidía completa (66.7%), seguido de euploidía (22.2%). La proporción de mosaicos fue de 5.6% y las alteraciones segmentales representaron solo el 2.2% del total de embriones analizados.
Por su parte, en el grupo secuenciación masiva más polimorfismos de nucleótido único, también predominó la aneuploidía completa (57.1%), aunque con una proporción menor que en NGS. Se observó un incremento en la detección de mosaicos (11.4%) y alteraciones segmentales (7.1%) respecto al grupo NGS. La tasa de euploidía fue similar entre ambos grupos (21.4% en SNP array vs 22.2% en NGS).
Tabla 1. Diagnóstico embrionario por secuenciación masiva versus secuenciación masiva con polimorfismos de nucleótido único.
Distribución de embriones clasificados como euploides, aneuploides, mosaicos, segmentales, de acuerdo con la técnica de diagnóstico genético preimplantacional empleada. Se presenta el porcentaje relativo dentro de cada grupo técnico (NGS o SNP-array). Este análisis evidencia una mayor proporción de euploides y detección de mosaicos y segmentales en el grupo evaluado con SNP-array, en comparación con secuenciación masiva únicamente.
Tabla 1. Diagnóstico embrionario por secuenciación masiva versus secuenciación masiva con polimorfismos de nucleótido único.
|
|
NGS |
SNP |
|
Euploide |
22.2% |
21.4% |
|
Aneuploide |
66.7% |
57.1% |
|
Mosaico |
5.6% |
11.4% |
|
Segmental |
2.2% |
7.1% |
La Figura 1 representa de manera comparativa la distribución diagnóstica de embriones analizados mediante secuenciación masiva y secuenciación masiva más polimorfismos de nucleótido único.
Se observa que en ambos grupos predomina la aneuploidía completa, aunque con una proporción mayor en el grupo NGS (60 embriones) frente al grupo SNP array (40 embriones).
Figura 1. Distribución diagnóstica comparativa
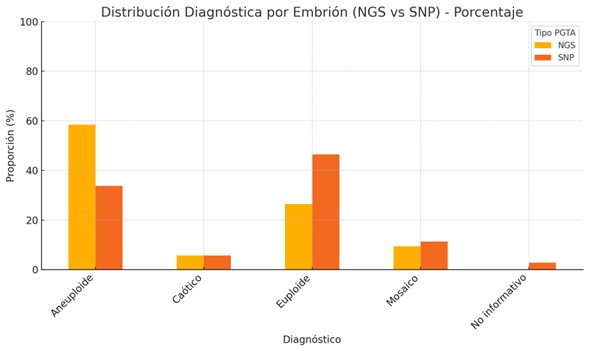
Figura 1. Distribución diagnóstica comparativa de embriones evaluados por secuenciación masiva versus secuenciación masiva con polimorfismo s de nucleótido único.
Se muestra la proporción de embriones clasificados como euploides, aneuploides, mosaicos y no informativos, de acuerdo con la técnica de análisis genético utilizada.
El análisis evidencia una mayor proporción de embriones euploides y detección de mosaicos en el grupo SNP-array, en contraste con la mayor prevalencia de aneuploides observada en el grupo NGS: NGS = Next Generation Sequencing; SNP = Single Nucleotide Polymorphism; PGTA = Preimplantation Genetic Testing for Aneuploidy.
La Tabla 2 presenta el análisis inferencial comparativo entre secuenciación masiva y secuenciación masiva más polimorfismos de nucleótido único, utilizando la prueba exacta de Fisher para evaluar la distribución de diagnósticos embrionarios entre ambas metodologías.
Tabla 2. Análisis inferencial de la distribución diagnóstica entre secuenciación masiva y secuenciación masiva con polimorfismo s de nucleótido único
Se compararon las proporciones de embriones clasificados como euploides, aneuploides, mosaicos y segmentales entre ambos métodos de diagnóstico genético preimplantacional. Aunque no se alcanzó significancia estadística clásica (p<0.05), se observaron tendencias clínicas relevantes, destacando una mayor detección de mosaicos y alteraciones segmentales en el grupo evaluado con SNP-array. El valor p reportado corresponde a la prueba de Fisher exact, seleccionada por la distribución de frecuencias observadas. NGS = Next-Generation Sequencing; SNP = Single Nucleotide Polymorphism; PGTA = Preimplantation Genetic Testing for Aneuploidy.
|
Clasificación |
p |
|
Euploide |
0.45 |
|
Aneuploidía |
0.62 |
|
Mosaico |
0.08 |
|
Segmental |
0.1 |
La representación gráfica de alteraciones cromosómicas (Figura 2) permitió visualizar las regiones cromosómicas más frecuentemente afectadas en la cohorte evaluada con secuenciación masiva combinada con polimorfismo s de nucleótido único-array.
Entre los hallazgos más relevantes destacan la frecuencia de alteraciones segmentales en cromosomas 1, 3, 4, 8, 14, 18, y 19, con localización recurrente en regiones p y q; la identificación de trisomías completas en cromosomas 2, 19, 20, 21, y 22, siendo la trisomía 21 una de las más recurrentes, la presencia de monosomía completa observada en cromosoma 21; pérdidas y ganancias segmentales en múltiples cromosomas, destacando la recurrencia en regiones 8p, 4p, y 14q.
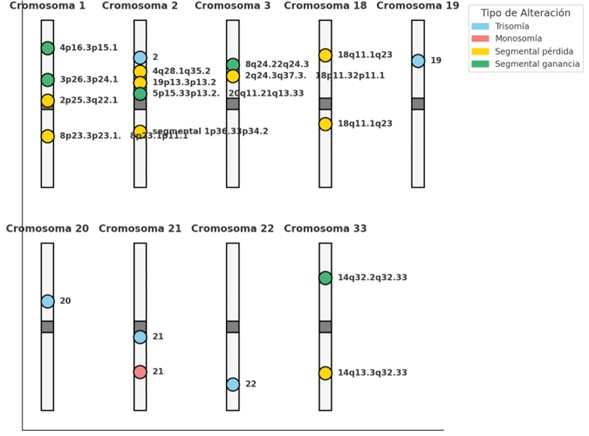
Figura 2. Representación gráfica de las alteraciones cromosómicas detectadas mediante secuenciación masiva con polimorfismo s de nucleótido único. Se muestran las alteraciones numéricas (trisomías y monosomías completas) y estructurales (ganancias y pérdidas segmentales). Las alteraciones fueron localizadas por color según el tipo de aberración: trisomía (azul), monosomía (rojo), ganancia segmental (verde) y pérdida segmental (amarillo). Las etiquetas indican la región cromosómica específica afectada.
Análisis de discordancia diagnóstica
Se realizó una comparación indirecta de distribuciones diagnósticas de embriones evaluados exclusivamente con NGS o con SNP-array.
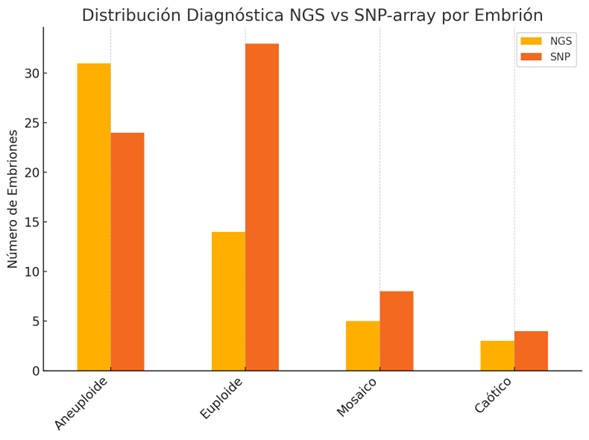
Figura 3. Distribución diagnóstica comparativa de embriones evaluados mediante secuenciación masiva versus PGTA. El gráfico presenta la proporción de embriones clasificados como euploides, aneuploides, mosaicos y caóticos.
El análisis inferencial mediante prueba de Chi-cuadrado mostró una tendencia hacia diferencias globales significativas (p = 0.0592), evidenciando una brecha diagnóstica clínicamente relevante entre ambas plataformas. NGS = Next Generation Sequencing; SNP = Single Nucleotide Polymorphism; PGTA = Preimplantation Genetic Testing for Aneuploidy.
La proporción de euploides fue significativamente mayor en el grupo evaluado con SNP-array (46.5%) respecto a NGS (26.4%). Por el contrario, la tasa de aneuploides fue considerablemente más alta con NGS (58.5%) comparado con SNP-array (33.8%). SNP-array también identificó una mayor frecuencia relativa de mosaicos (11.3%) y segmentales (no discriminados en la tabla, pero observados previamente), además de detectar casos de diagnóstico no informativo (2.8%), los cuales no fueron reportados en el grupo NGS. Aunque la prueba de chi-cuadrado mostró una tendencia hacia diferencias significativas (p = 0.0592), no se alcanzó el umbral de significancia estadística (p<0.05) (Figura 3).
Algoritmo clinico
Con base en estos hallazgos, se diseñó un algoritmo clínico preliminar (Figura 4), que representa las implicaciones prácticas de la discordancia diagnóstica en la toma de decisiones clínicas. El algoritmo parte del grupo general de embriones candidatos a diagnóstico genético preimplantacional, diferenciando dos rutas según la técnica empleada:
- secuenciación masiva: Se asocia con mayor tasa de aneuploidías, menor detección de euploides y baja identificación de mosaicos/segmentales.
- secuenciación masiva combinada con polimorfismo s de nucleótido único-array: Muestra mayor tasa de euploides, mejor detección de mosaicos/segmentales y un perfil diagnóstico más completo.
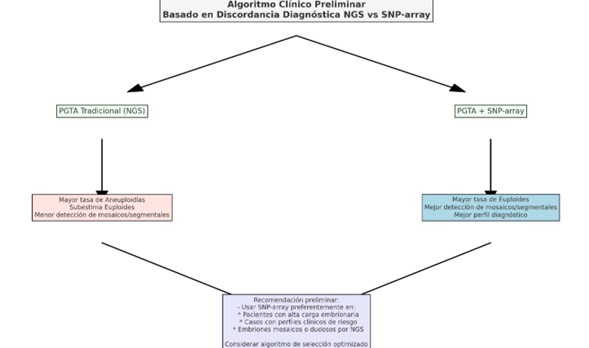
Figura 4. Algoritmo clínico preliminar para la integración de secuenciación masiva y secuenciación masiva con polimorfismo s de nucleótido único en la selección embrionaria personalizada. El esquema representa la propuesta de flujo de decisión basado en la discordancia diagnóstica observada entre ambas plataformas, diferenciando rutas según la técnica empleada. La ruta de secuenciación masiva se asocia a una mayor proporción de embriones aneuploides y menor detección de mosaicos y segmentales, mientras que la combinación con SNP-array permite una caracterización genética más completa, con mayor proporción de euploides y detección optimizada de alteraciones estructurales y mosaicos. NGS = Next-Generation Sequencing; SNP = Single Nucleotide Polymorphism; PGTA = Preimplantation Genetic Testing for Aneuploidy.
Discusión
En la presente serie clínica, el análisis comparativo de la distribución diagnóstica obtenida mediante secuenciación masiva versus secuenciación masiva con polimorfismo s de nucleótido único evidenció una brecha diagnóstica clínicamente relevante, con una mayor proporción de embriones euploides (46.5% vs 26.4%) y menor tasa de aneuploidías (33.8% vs 58.5%) en el grupo evaluado con polimorfismos de nucleótido único.
EEstos hallazgos son consistentes con estudios que han demostrado que el uso de plataformas basadas en SNP-array combinadas con secuenciación de última generación mejora la resolución diagnóstica, permitiendo una mejor detección de alteraciones segmentales submicroscópicas, mosaicos de bajo grado y anomalías estructurales, elementos que pueden ser subestimados en análisis realizados únicamente mediante secuenciación masiva únicamente 6,7.
En particular, Munné et al.6 reportaron que la integración de SNP-array permitió reclasificar aproximadamente el 10-15% de los embriones inicialmente considerados aneuploides por secuenciación masiva, mejorando la identificación de embriones potencialmente viables.
Del mismo modo, el estudio de Kushnir et al. 8 enfatizó que el uso combinado de secuenciación de última generación combinada con polimorfismos de nucleótido único aumenta la detección de alteraciones segmentales hasta en un 50% respecto a secuenciación masiva solamente, particularmente en regiones cromosómicas susceptibles como 8p, 14q y 18q.
Estas diferencias se atribuyen a la mayor densidad de marcadores y la capacidad del polimorfismos de nucleótido único de evaluar tanto el número de copias como los patrones de heterocigosidad, lo que permite la detección de aberraciones estructurales subclínicas no identificadas por secuenciación masiva.8
Aunque la diferencia en tasas de euploides no alcanzó significancia estadística estricta en nuestro análisis (p=0.0592), la tendencia observada coincide con lo reportado por Franasiak et al. (2014), quienes sugieren que la sobreestimación de aneuploidías por secuenciación de última generación puede llevar a una subestimación clínica de embriones potencialmente transferibles, con el consecuente impacto en la optimización de tasas de embarazo y reducción de descartes innecesarios.
Es evidente que, aunque la aneuploidía completa continúa siendo el diagnóstico predominante en la población evaluada, las alteraciones de tipo mosaico y segmentales juntas representan aproximadamente el 18.5% de los diagnósticos.
Este hallazgo refuerza la importancia clínica del uso de polimorfismos de nucleótido único como herramienta complementaria en el diagnóstico genético preimplantacional, permitiendo la detección de alteraciones estructurales y mosaicos de bajo grado que podrían pasar inadvertidos en metodologías tradicionales, optimizando así la toma de decisiones clínicas individualizadas y la selección embrionaria en pacientes de mayor riesgo.
Aunque ninguna categoría alcanzó diferencias estadísticamente significativas (p > 0.05), se observó una tendencia clínica relevante en la detección de mosaicos (p = 0.08) y segmentales (p = 0.10), sugiriendo un potencial incremento en la sensibilidad diagnóstica del secuenciación masiva más polimorfismos de nucleótido único frente a la metodología de secuenciación masiva, especialmente en la identificación de estas alteraciones de difícil diagnóstico.
Estas tendencias, aunque limitadas por el tamaño muestral, respaldan la necesidad de validar estos hallazgos en estudios prospectivos de mayor escala, así como explorar la implementación dirigida de polimorfismos de nucleótido único array en perfiles clínicos específicos que podrían beneficiarse de una mayor resolución diagnóstica.
Los hallazgos reflejan la alta incidencia de alteraciones segmentales y numéricas en cromosomas críticos como 8, 14, 18, y 21, consistentes con reportes previos en literatura que destacan la vulnerabilidad de estas regiones en el desarrollo embrionario temprano. La presencia de alteraciones segmentales pequeñas en regiones como 8p23.3 y 14q32, no detectables mediante secuenciación masiva, enfatiza la ventaja diagnóstica que ofrece la combinación con polimorfismos de nucleótido único, permitiendo una detección más fina y precisa de aberraciones estructurales.
La identificación recurrente de trisomía 21 y alteraciones segmentales en cromosomas 8 y 14 observada en nuestro análisis es coherente con estudios previos que documentan la alta susceptibilidad de estas regiones a errores de segregación durante la meiosis y mitosis temprana 9,10
En particular, el estudio de Kushnir et al. 8 reportó que el uso de SNP-array de alta resolución permitió identificar frecuencias aumentadas de pérdidas y ganancias segmentales en 8p23.3, 14q32 y 18q11, hallazgos que también se reflejan en nuestra cohorte, consolidando la evidencia de que estas regiones representan puntos importantes de inestabilidad cromosómica embrionaria. Además, la capacidad de detectar alteraciones segmentales pequeñas (<10 Mb), como las observadas en nuestra representación, ha sido señalada como una de las principales ventajas diagnósticas del SNP-array frente a secuenciación masiva únicamente 10, 11
La integración de este ideograma clínico aporta un valor adicional a la interpretación clínica, ya que permite correlacionar visualmente la carga cromosómica embrionaria con los perfiles clínicos, apoyando la toma de decisiones reproductivas personalizadas 12
Desde una perspectiva clínica, la representación utilizada facilita la comunicación gráfica con equipos médicos y pacientes, mejorando la comprensión del tipo y distribución de las alteraciones identificadas. De igual manera, este enfoque podría ser extrapolado como una herramienta estandarizada de perfil genético cromosómico embrionario, integrándose en algoritmos de toma de decisiones reproductivas personalizadas.
Se recomienda ampliar este análisis en cohortes más grandes y correlacionar las alteraciones cromosómicas detectadas con los resultados clínicos reproductivos y obstétricos, para validar su valor predictivo y su utilidad en la práctica clínica cotidiana.
Encontramos que el estudio por polimorfismos de nuclétido único presenta una mayor proporción de euploides y menor proporción de aneuploides en comparación con secuenciación ,asiva, como ya se había observado gráficamente.Aunque la diferencia global no alcanza significancia estadística estricta, la magnitud de la brecha clínica es relevante y consistente con la hipótesis de que los polimorfismos de nuclótido único reduce la sobreestimación de aneuploidías observada con secuenciación masiva.
El análisis inferencial mediante Chi-cuadrado aplicado a la distribución diagnóstica por técnica (secuenciación masiva vs secuenciación masiva combinada con polimorfismos de nucleótido único) evidenció una tendencia hacia diferencias globales significativas (p = 0.0592), indicando una brecha diagnóstica potencialmente relevante desde el punto de vista clínico.
Estos resultados son consistentes con lo reportado en la literatura, donde diversos estudios han documentado que la adición de polimorfismos de nucleótido único a las plataformas de secuenciación masiva mejora la resolución diagnóstica y reduce la probabilidad de falsos positivos de aneuploidía, especialmente en la detección de segmentales pequeños y mosaicos intermedios (Kushnir et al., 2022; Marin et al., 2020).
En particular, Kushnir et al. (2022) mostraron que la detección de alteraciones segmentales se incrementa significativamente con polimorfismos de nucleótido único, alcanzando regiones de hasta 5 Mb, no identificables mediante secuenciación masiva únicamente, lo que impacta de manera directa en la selección embrionaria clínica.
Por otra parte, Treff et al. (2016) demostraron que los polimorfismos de nucleótido único ofrece una mayor consistencia diagnóstica en la detección de mosaicos y aneuploidías parciales, minimizando la variabilidad intra-técnica observada en secuenciación masiva, y mejorando la confianza clínica en la clasificación de embriones viables
Cabe destacar que la falta de significancia estadística puede explicarse, en parte, por el tamaño muestral limitado y la heterogeneidad clínica de la cohorte analizada, limitaciones que también han sido reconocidas en estudios previos (Marin et al., 2020; Kushnir et al., 2022).
No obstante, la tendencia consistente a una mejor discriminación diagnóstica por polimorfismos de nucleótido único sugiere que futuros estudios con muestras ampliadas y seguimiento clínico podrían confirmar la relevancia estadística de estas diferencias observadas, consolidando la propuesta de algoritmos de selección embrionaria basados en perfiles de riesgo integrados y plataformas diagnósticas combinadas.
El algoritmo clínico preliminar corregido propuesto en este estudio integra de manera estratégica los hallazgos derivados de la discordancia diagnóstica observada entre secuenciación masiva y secuenciación masiva con polimorfismo s de nucleótido único, planteando una ruta clínica diferenciada y personalizada para la toma de decisiones reproductivas.
La representación visual del algoritmo fue diseñada con nodos diferenciados y flechas de flujo, facilitando la comprensión del impacto clínico de las discrepancias diagnósticas observadas, así como su aplicabilidad práctica en el contexto de la toma de decisiones clínicas reproductivas.
El algoritmo planteado en el presente estudio destaca que en escenarios clínicos convencionales o con baja carga embrionaria, la evaluación mediante secuenciación masiva únicamente podría ser suficiente como primera línea diagnóstica. Sin embargo, en pacientes con alta carga embrionaria, hallazgos diagnósticos ambiguos, presencia de mosaicos bajos o sospecha de alteraciones segmentales, se propone integrar polimorfismos de nucleótido único como herramienta diagnóstica complementaria, con el objetivo de refinar la clasificación embrionaria y reducir la tasa de falsos positivos de aneuploidía (Munné et al., 2019; Marin et al., 2020).
Esta recomendación se fundamenta en la evidencia reportada por Treff et al. (2016), quienes demostraron que el uso de polimorfismos de nucleótido único mejora la consistencia diagnóstica y disminuye la probabilidad de error diagnóstico, especialmente en la detección de mosaicos y aneuploidías segmentales, lo que impacta de manera directa en las tasas de transferencia y embarazo clínico. Asimismo, el estudio de Kushnir et al. (2022) refuerza que la incorporación de esta técnica aporta una sensibilidad diagnóstica superior para alteraciones estructurales, recomendando su integración dirigida en pacientes con perfiles de riesgo genético elevado.
Aunque este algoritmo preliminar requiere validación en estudios multicéntricos y con seguimiento clínico de resultados reproductivos, su diseño es congruente con las tendencias emergentes de medicina embrionaria personalizada, que abogan por modelos de diagnóstico y selección basados en la integración de plataformas genómicas complementarias y datos fenotípicos individuales (Franasiak et al., 2014).
El presente estudio indica que secuenciación masiva más polimorfismos de nucleótido único mostró una tendencia a incrementar la detección de mosaicos de bajo grado y alteraciones segmentales en comparación con secuenciación masiva, aun cuando estas diferencias no alcanzaron significancia estadística, probablemente debido al tamaño limitado de la muestra. Estos hallazgos son consistentes con la literatura que señala una mayor resolución diagnóstica de los polimorfismos de nucleótido único para identificar alteraciones cromosómicas estructurales y mosaicos sutiles, contribuyendo a una selección embrionaria más precisa y personalizada. Las limitaciones incluyen el diseño retrospectivo, el tamaño de muestra y la falta de correlación clínica de los hallazgos.
Conclusiones
La integración de polimorfismos de nucleótido único a la evaluación embrionaria mediante diagnóstico genético preimplantacionalamplía de manera significativa la capacidad diagnóstica, permitiendo una mejor detección de alteraciones segmentales, mosaicos y reduciendo la tasa de falsos positivos de aneuploidía observados en secuenciación masiva únicamente.
La discordancia diagnóstica identificada entre secuenciación masiva únicamente y polimorfismos de nucleótido único evidencia una brecha clínica relevante, particularmente en la clasificación de embriones euploides y mosaicos, con implicaciones directas en la selección embrionaria y en los resultados reproductivos.
El análisis inferencial, aunque limitado por el tamaño muestral, mostró tendencias consistentes que refuerzan la necesidad de considerar la integración de plataformas complementarias para una caracterización genética embrionaria más precisa, especialmente en pacientes con perfiles de riesgo.
El algoritmo clínico preliminar propuesto plantea una estrategia personalizada para la aplicación de Polimorfismos de nucleótido único, sugiriendo su uso dirigido en escenarios de incertidumbre diagnóstica o riesgo clínico elevado, optimizando la toma de decisiones en medicina reproductiva.
Futuros estudios con cohortes más amplias y seguimiento clínico serán necesarios para validar estos hallazgos y definir con mayor precisión el impacto de la integración de polimorfismo de nucleótido único en las tasas de éxito reproductivo y en la eficiencia de la selección embrionaria personalizada.
Bibliografía
- Practice Committee of the American Society for Reproductive Medicine. The use of preimplantation genetic testing for aneuploidy: a committee opinion. Fertil Steril. 2018;109(3):429-436.
- Harper JC, SenGupta SB. Preimplantation genetic diagnosis: state of the art 2011. Hum Genet. 2012;131(2):175-186.
- HFEA. Pre-implantation genetic testing for aneuploidy (PGT-A). Human Fertilisation and Embryology Authority. https://www.hfea.gov.uk/treatments/treatment-add-ons/pre-implantation-genetic-testing-for-aneuploidy-pgt-a/
- Munné S, Wells D. Detection of mosaicism at blastocyst stage with the use of high-resolution next-generation sequencing. Fertil Steril. 2017;107(5):1085-1091.
- Treff NR, Zimmerman RS. Advances in preimplantation genetic testing for aneuploidy using next-generation sequencing. Fertil Steril. 2017;107(3):593-599.
- Victor AR, Tyndall JC, Brake AJ, et al. One hundred mosaic embryos transferred prospectively in a single clinic: exploring when and why they result in healthy pregnancies. Fertil Steril. 2019;111(2):280-293.
- Munné S, Kaplan B, Frattarelli JL, Child T, Nakhuda G, Shamma FN, Silverberg K, Kalista T, Handyside AH, Katz-Jaffe M, Wells D, Gordon T, Stock-Myer S, Willman S; STAR Study Group. Preimplantation genetic testing for aneuploidy versus morphology as selection criteria for single frozen-thawed embryo transfer in good-prognosis patients: a multicenter randomized clinical trial. Fertil Steril. 2019 Dec;112(6):1071-1079.e7. doi: 10.1016/j.fertnstert.2019.07.1346. Epub 2019 Sep 21. PMID: 31551155.
- Kushnir VA, Darmon SK, Shapiro JL, et al. Novel insights into the detection of segmental aneuploidies using high-resolution SNP microarrays in human blastocysts. Hum Reprod. 2022;37(2):298-307. doi:10.1093/humrep/deab244
- Franasiak JM, Forman EJ, Hong KH, et al. The nature of aneuploidy with increasing age of the female partner: a review of 15,169 consecutive trophectoderm biopsies evaluated with comprehensive chromosomal screening. Fertil Steril. 2014;101(3):656-663. doi:10.1016/j.fertnstert.2013.11.004
- Marin D, et al. Comprehensive chromosome screening and its clinical impact: high-resolution SNP-array improves diagnosis of segmental and mosaic aneuploidies. Fertil Steril. 2020;113(5):1078-1086. doi:10.1016/j.fertnstert.2020.01.018
- Treff NR, Eccles J, Marin D, et al. SNP microarray-based 24 chromosome aneuploidy screening is significantly more consistent than FISH. Mol Hum Reprod. 2016;22(10):744-749. doi:10.1093/molehr/gaw049
- Capalbo A, et al. Clinical validation of a targeted next-generation sequencing-based comprehensive chromosome screening platform in a prospective, randomized, blinded, multicenter study. Fertil Steril. 2021;116(5):1223-1230. doi:10.1016/j.fertnstert.2021.06.008